알파폴드2 모델 분석
알파폴드2의 작동 원리
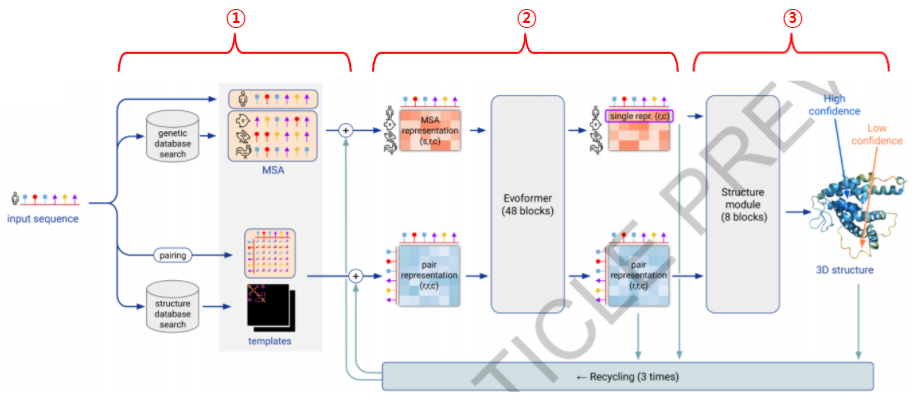
이 그림은 알파폴드2의 처리 과정을 한눈에 보여줍니다. 알파폴드2는 다음의 3가지 스텝으로 구성되어 있습니다. 먼저 입력 데이터를 전처리하는 Input feature embeddings(①) 단계, 어텐션 학습을 통해 전처리된 데이터에서 필요한 정보를 뽑아내는 Evoformer(②)단계, 그리고 여기서 나온 정보를 구체적인 3차원 좌표로 처리하는 Structure module(③)단계입니다.
1. Input feature embeddings
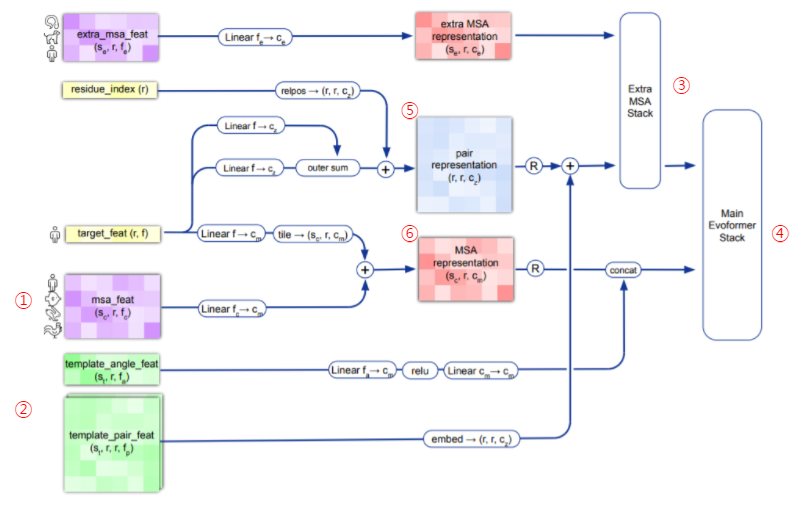
알파폴드2의 첫 단계는 알파폴드1과 마찬가지로 입력 시퀀스를 전처리 하는 단계입니다. 먼저 유전자 데이터베이스에서 유사한 evolutionary 서열을 검색하여 다중 서열 정렬(MSA)을 생성합니다(①). 170,000개의 PDB 데이터, Uniprot의 대규모 데이터베이스를 이용했으며, JackHMMER와 HHblits를 이용해서 UniRef90, MGnify clusters, BFD를 검색하는 방식을 썼습니다.
알파폴드2는 여기에 추가로 쿼리 시퀀스와 유사한 시퀀스 부분을 가진, 알려진 단백질 템플릿을 검색합니다(②). 알파폴드2가 기존의 Yang Zhang 랩이나 David Baker랩을 뛰어 넘을 수 있었던 건 시퀀스 정보에서 필요한 정보를 추가하는 Extra MSA Stack(③), Evoformer Stack(④)과정이 있다는 것입니다.
2. Evoformer
Pair representation과 MSA representation이 만들어지면 이 정보는 Evoformer단계로 넘어갑니다.
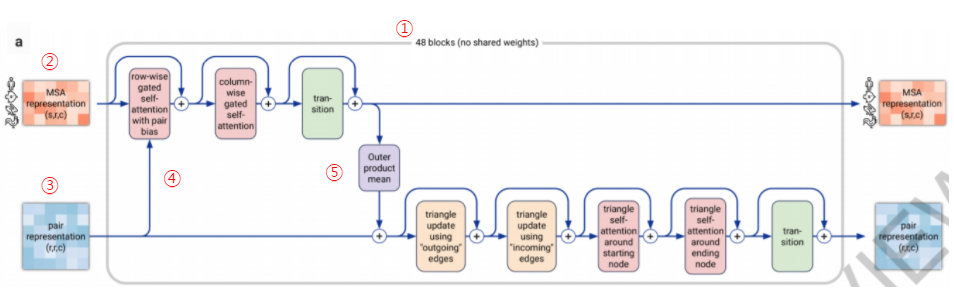
주어진 Pair representation과 MSA representation을 개선하기 위해 어텐션 메커니즘의 48개 레이어로 구성된 deep tranformer-like 네트워크(①)가 적용되는 단계입니다. 48개의 모든 레이어에는 각각 개별 매개변수가 있으며 입력과 출력은 MSA representation과 Pair representation입니다.
Evoformer 안에는 두개의 흐름이 있는데, 하나는 MSA representation이 입력되어 진행되는 흐름으로 위쪽의 흐름입니다(②). 또 하나는 Pair representation(③)의 정보 흐름입니다. 이 두 흐름의 아이디어는 기본적으로 단백질의 공간적, 진화적 관계에 대한 직접적인 추론을 가능하게 하는 정보를 교환하게 하는 것입니다.
3. Structure module
Evoformer단계에서 만들어진 정보는 protein geometry의 구체적인 3차원 좌표로 변환하는 Structure module단계로 넘어옵니다.
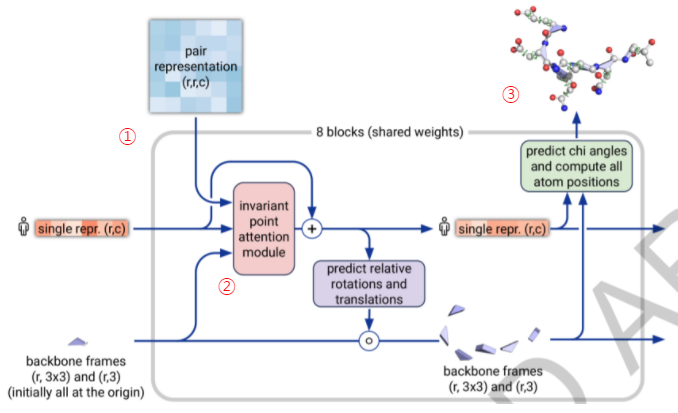
Evoformer가 가지고 있는 정보는 2D representation 형태로, 반드시 3차원 단백질 기하학으로 변환되어야 합니다. 이는 weight을 공유하는 8개의 RNN 블록에서 수행됩니다. Evoformer의 최종 MSA representation 정보와 Pair representation 정보가 사용되며, distances, torsions, atom coordinates, Cα-lDDT의 추정치를 예측하게 됩니다.
Invariant Point Attention 메커니즘에는 단백질 구조 개선을 위해 특별히 설계된 여러가지 기술이 포함되어 있습니다. 이중 하나가 시퀀스 표현과 백본 변환을 반복적으로 업데이트함으로써 단백질의 블랙홀 초기화가 최종 백본 기하학이 나타날 때까지 점진적으로 조정되게 하는 것입니다.
마치며
알파폴드는 CASP14에서 0부터 100까지의 범위를 가지는 GDT점수(100이 만점)에서 median score 92.4점을 얻었습니다. 이는 약 1.6 Angstroms의 평균 오차(RMSD)를 가진다는 의미인데, 곧 원자의 너비 (0.1 나노 미터)와 비슷한 수준입니다.
결론적으로 Attention-based neural network을 통해 End-to-end 방식으로 학습했다고 할 수 있으나, 이러한 간단한 설명으로는 턱없이 부족할 만큼 획기적인 아이디어들이 적재 적소에 사용된 혁신적인 시스템이라는 생각입니다.
1972년 아미노산 배열의 1차 구조가 처음으로 해석된 이래, 단백질의 구조를 알아내기 위한 노력이 계속되어 왔습니다. 알파폴드2는 지난 50여년간 이어온 분자 생물학, 유전학, 생명정보학의 성과가 AI의 급속한 발전과 더불어 탄생한 놀라운 사건이 아닐 수 없습니다.
2021년 7월
알파폴드 리뷰와 해석
알파폴드와 CASP
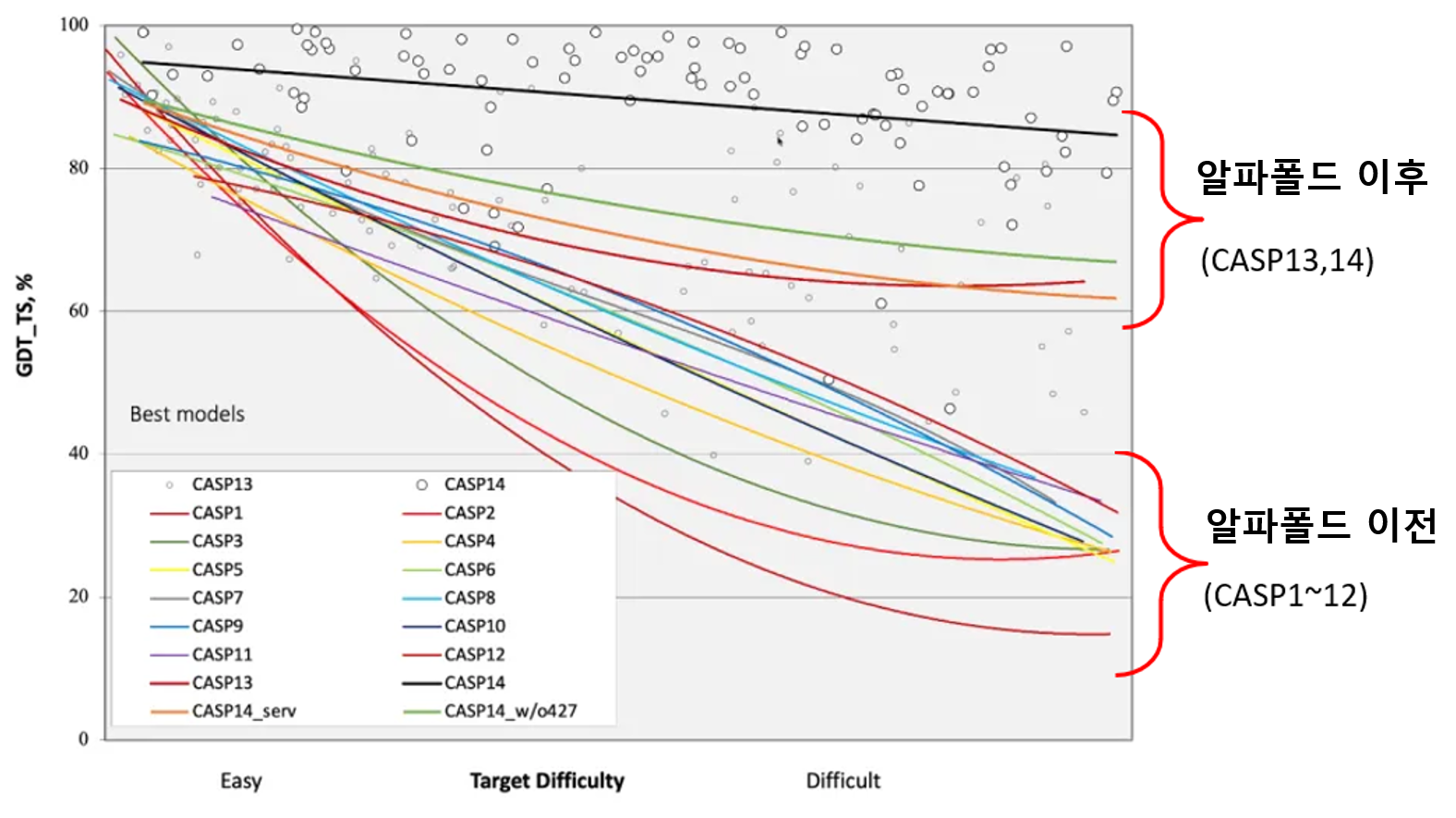
흥미로운 그림 하나를 가지고 왔습니다. 2020년 1월, PNAS에 실린 데이빗 베이커 그룹 논문의 결과를 설명하는 그림입니다. c부분을 보면 주황색으로 표시된 막대가 알파폴드보다 앞서 있는 것을 알 수 있습니다. 이들이 만든 trRosetta가 알파폴드를 뛰어 넘었다는 뜻입니다.
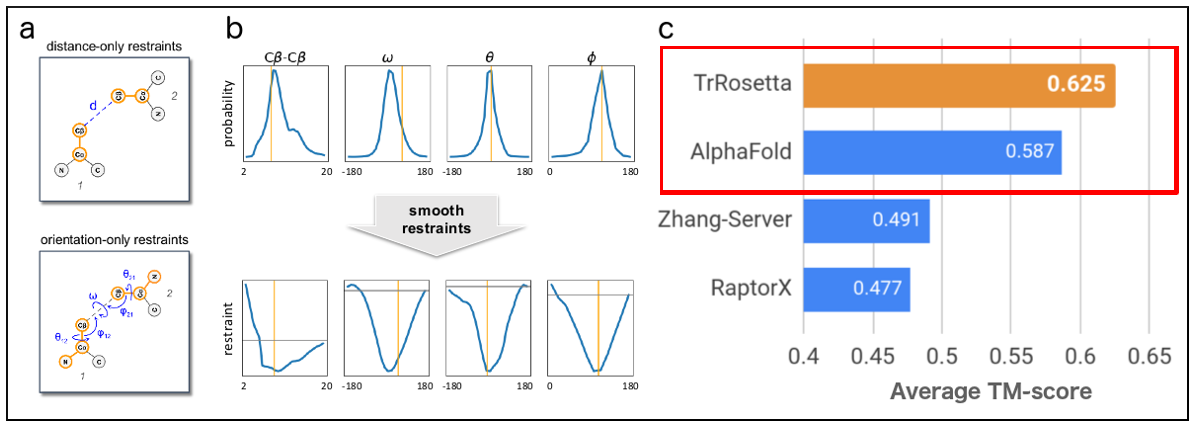
흥미를 더할 만한 포인트가 몇개 더 있습니다. 2018년, 제13회 단백질 구조 예측 대회(CASP13)에 처음 등장 해 기존의 탑 그룹 결과물들을 월등한 차이로 이긴 바 있는 알파폴드가, 탑 그룹 중에서도 큰 형 격인 워싱턴대학교 베이커 교수 그룹에 의해 단 1년여 만에 따라 잡혔다는 뜻이기도 합니다. 베이커 그룹은 한발 더 나아가 trRosetta를 누구나 쓸 수 있도록 했습니다. 그러면 다가올 제14회 단백질 구조 예측 대회의 결과에도 시선이 쏠릴 수밖에 없습니다.
2020년 12월, 기다리던 제14회 단백질 구조 예측대회의 뚜껑이 열렸습니다. 그리고 사람들은 다시 한번 충격에 빠집니다.
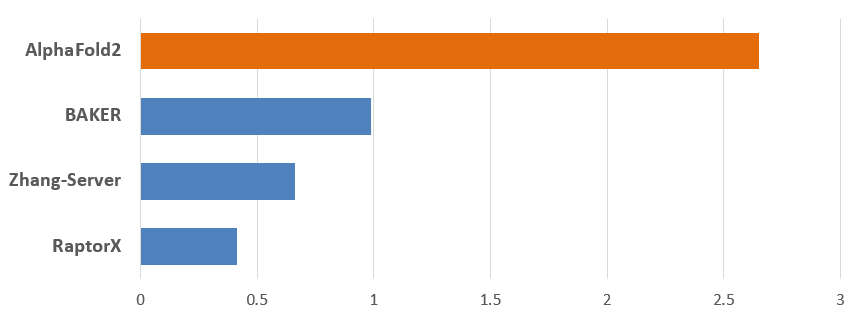
완패입니다. 알파폴드2를 앞세워 참여한 딥마인드의 결과를 추월하기는 커녕 격차가 더 벌어 졌습니다. 이상하군요. 분명 베이커 그룹의 trRotessta는 딥마인드의 알파폴드를 앞섰었습니다. 아무리 알파폴드의 버전이 달라 졌다고 해도, 베이커 그룹의 내공도 만만치 않았을 것입니다. 그럼에도 이토록 큰 성능의 차이라니, 무언가 놓치는 부분이 있는 걸까요? 1972년 노벨상을 받으며 시작한 50여년 학계의 노하우가 이토록 크게 뒤쳐지는 이유, 그들이 결코 알파폴드를 따라잡을 수 없었던 이유에 대해서 지금부터 하나씩 풀어 보고자 합니다.
알파폴드의 원리
2018년, 단백질 구조 예측 대회에 처음 모습을 드러낸 딥마인드의 알파폴드는 모든 참가자들을 놀라게 하기에 충분했습니다. 26년째 개최되어온 단백질 구조 예측 대회에 처음 참가한 그들이 지난 수십년 간의 학문적 성과를 오롯이 담은 기존의 단백질 구조 예측 툴들을 단숨에 앞질렀기 때문입니다.
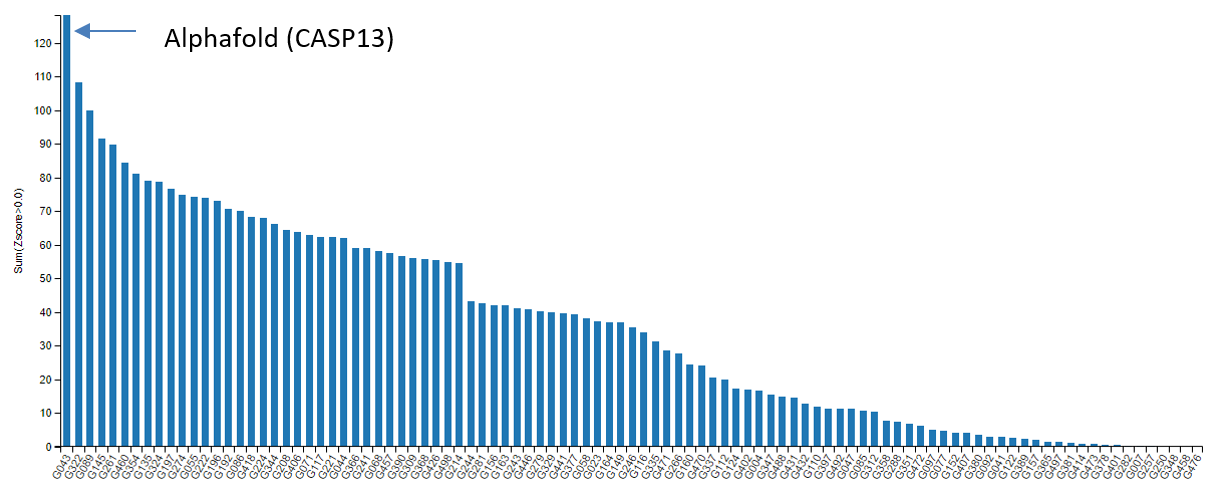
사람들은 그들이 어떻게 했는지 궁금해했습니다. 그들의 발표에 귀를 기울였고, 논문을 기다렸습니다. 딥마인드는 자신의 노하우를 꽁꽁 숨겨두지 않았습니다. 두번의 논문을 발표했고 소스 코드를 공개해 자신들의 방법에 대한 재현의 길을 터 주었습니다.
미지의 구조를 예측하기 위한 알파폴드의 접근 방법은 다음 그림 한장으로 요약됩니다.
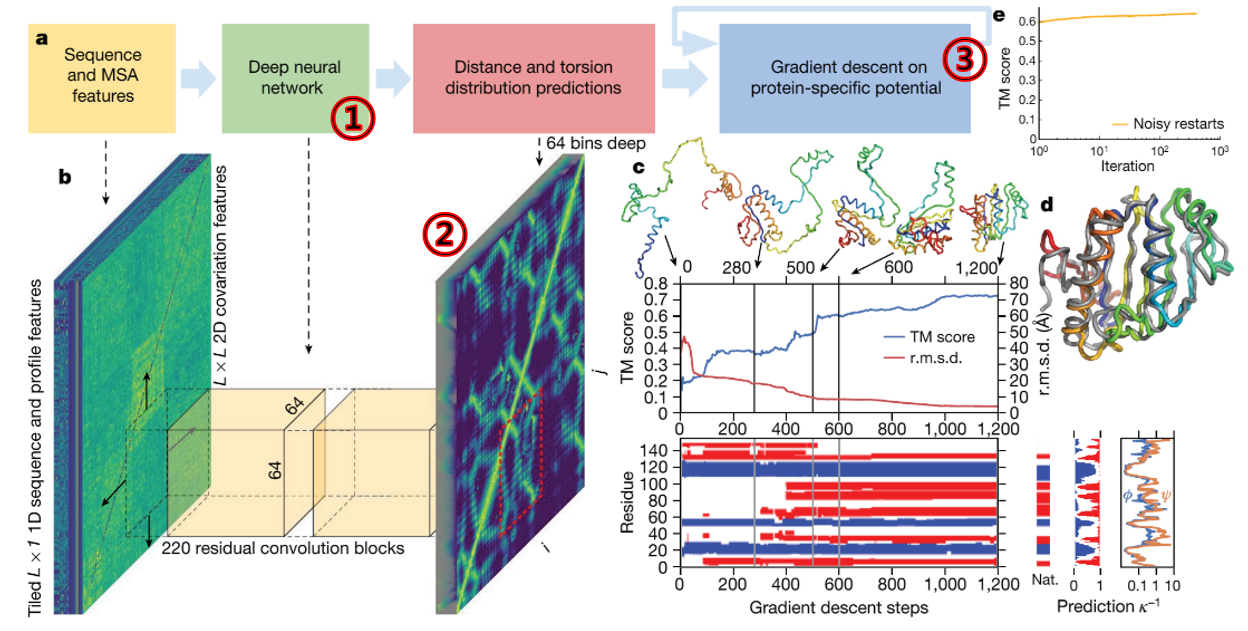
주목해야 하는 세가지 키워드는, 딥러닝(①), Distance Map(②) 그리고 경사하강법(③)입니다. 딥러닝(①)을 위해서 다중 서열 정렬 (MSA)을 이용했고, 학습 알고리즘으로 컨볼루션 뉴럴 네트웍(CNN)을 썼으며 그 결과는 Distance Map(②)으로 나타납니다.
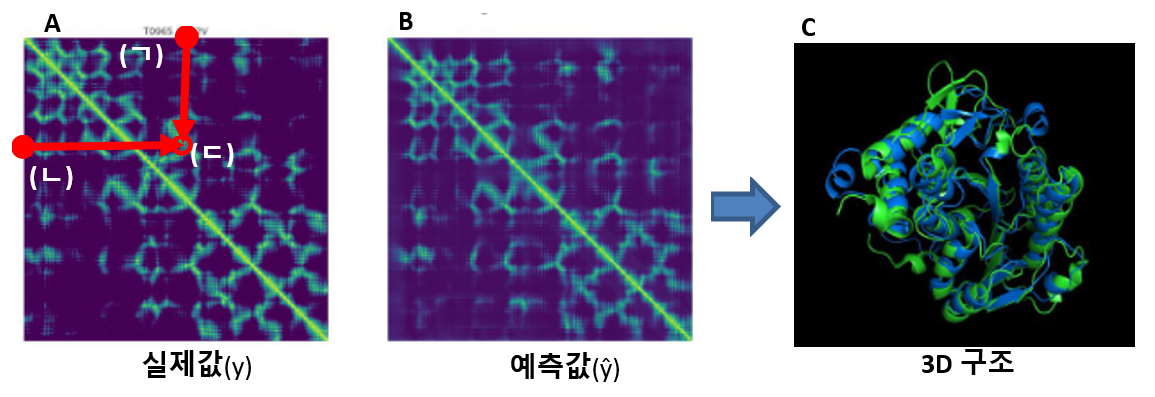
A는 단백질의 실제 3차원 구조를 이용해 만든 Distance Map입니다. B는 컨볼루션 과정을 거쳐 예측된 Distance Map입니다. 이 A, B의 가로축과 세로축은 단백질을 이루고 있는 아미노산 서열들입니다. 두 축의 값이 서로 만나는 지점은 배열사이의 거리를 나타냅니다. 단백질 3차원 입체 구조는 이렇게 해서 2차원 그림으로 표현되고, 이렇게 예측된 B그림이 실제 A그림과 유사해지도록 경사하강법으로 오차를 조절하는 것이 바로 알파폴드의 핵심입니다.
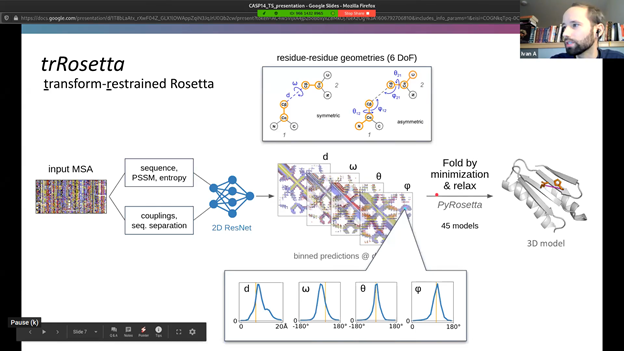
trRosseta개발 그룹도 이 눈문을 보면서 똑같이 분석했을 것입니다. 그리고 알파폴드에 보충할 만한 아이디어를 찾아보기 시작했을 것입니다. 베이커 그룹이 한가지 아이디어를 냈습니다. 잠깐, 왜 두 배열 사이의 ‘거리’만 보는 거지? 두 배열 사이의 ‘각도’를 고려하면 더 나은 결과가 나오지 않을까? 이 아이디어는 세계 최고의 실력자들로 이루어진 베이커 그룹의 연구원들에 의해 멋지게 실현되었고 알파폴드의 기본 결과물에서 한단계 업그레이드된 결과를 내 놓습니다. 이렇게 해서 나온 결과가 바로 “알파폴드를 뛰어 넘은 툴”, trRosetta였습니다.
따라잡지 못한 이유
하지만 이미 전술했듯, 다시 맞붙은 다음 대회에서 trRosetta를 앞세운 베이커 그룹의 결과는 알파폴드2에 크게 뒤쳐지고 맙니다. 베이커 그룹이 아무것도 잘 못하지 않았다는 것이 중요합니다. 베이커 그룹은 자신의 이전 대회 성적을 거뜬히 앞섰고, 자신이 그동안 보여준 모든 예측 결과보다도 좋은 결과를 선보였습니다. 그럼에도 불구하고 알파폴드와 너무나 큰 격차가 난 이유는 무엇일까요.
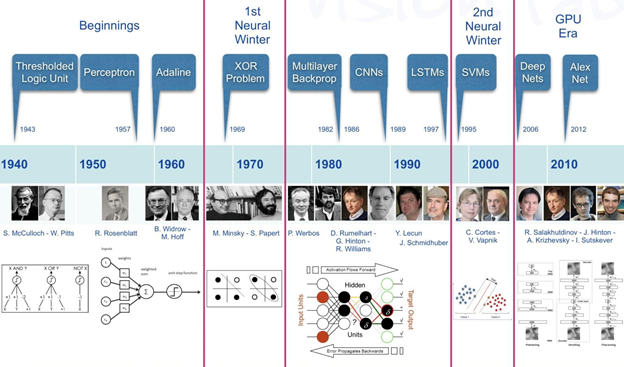
알파폴드의 탄생도 어쩌면 비슷합니다. 1943년, 신경과학자 워렌 맥컬럭이 허드렛일을 하던 월터 피츠의 능력을 알아보고 그와 함께 2진법 논리회로 모델을 만든 결과는 1960년대의 퍼셉트론을 지나, 역전파를 재발견하고 딥러닝의 기초를 쌓는 제프리 힌튼에 이르러 지금의 AI, 딥러닝이 됩니다.
다른 한편에선 아미노산 배열의 1차 구조를 처음으로 해석한 크리스천 안핀슨이 1972년 노벨상을 받은 이후 X-선 결정학, 핵 자기 공명, 극저온 전자 현미경과 같은 실험 기술의 발달이 이어졌고, 분자 생물학, 유전학, 생명 정보학의 성과는 대규모 데이터 축척의 시대를 맞이하게 했습니다. 이 거대한 두 흐름이 맞물린 2018년, 알파폴드가 탄생한 것입니다.
다시 질문으로 돌아가 보겠습니다. 학계는 최선을 다해 자신의 영역안에서 알파폴드의 부족한 점을 채웠는데 왜 따라잡지 못했는가? 어쩌면 답은 질문 안에 있을지도 모릅니다. 학계가 최선을 다해 자신의 영역을 고수했다는 것은 결국 학계는 최선을 다해 자신의 영역에 머물렀다는 뜻이기 때문입니다.
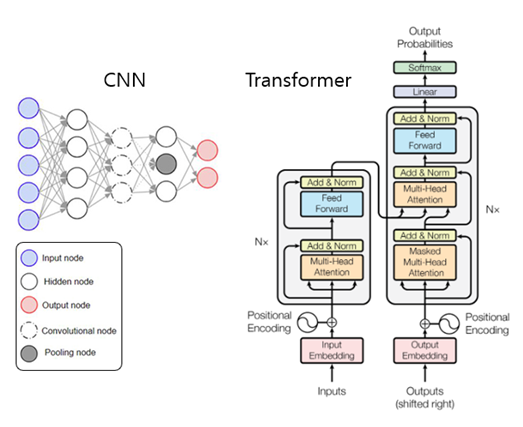
학문의 특성상 화학적, 생물학적 지식은 짧은 시기에 “폭발적”으로 발전할 만한 것이 아닙니다. AI와 다른 점입니다. 다시 말해, Cβ-Cβ의 Distance에 d, ω, θ, φ각도를 더하는 베이커 그룹의 노력은, CNN을 과감히 버리고 AI의 흐름에 발맞추어 Transformer와 Attention을 도입한 알파폴드2를 결코 따라잡을 수 없다는 것입니다.
활짝 열린 문
시대가 바뀌고 있고 세상이 변하고 있습니다. 학문의 경계 안에서 그동안 평가의 잣대로 사용되던 기준이 변하고 있고 나의 영역을 고수하는 것이 오히려 나를 그 안에 가두어 놓는 시대를 살고 있습니다. 이제 내 영역 안에 딥러닝이라는 툴을 가져와 쓰는 것을 넘어, 딥러닝을 제대로 알고 AI를 공부해서 기존의 영역을 다시 점검해야 할 때가 찾아왔습니다.
아직 기회는 문을 열어 놓고 우리를 기다리고 있습니다. 알파폴드는 결코 뛰어난 사람들이 천재적인 기술로 만든, 범접할 수 없는 소프트웨어가 아닙니다. 여전히 AI시대의 초입이라는 건 좋은 소식입니다. 지금은 딥러닝을 쉽게 배울 수 있는 때입니다. 필요한 대부분이 라이브러리로 준비되어 있고, 깃허브에는 잘된 소스코드가 공유되어 있습니다. 연구와 학문의 길을 걷는 사람이라면, 자신의 분야에 그동안 쏟아 부은 에너지의 작은 부분만으로도 AI라는 거인의 어깨에 올라탈 수 있습니다.
2021년 3월
© 2025 Taeho Jo. All rights reserved.

